Статья в IEEE Spectrum освещает проблему, о которой, по моему мнению, мало кто из нас знает. Простой факт заключается в том, что генетика геномика данные с такой скоростью, что без усовершенствованных алгоритмов сжатия эта тема не оправдает возложенных на нее надежд.
Прежде чем перейти к серьезным вещам, интересно отметить, что авторы статьи, Дмитрий Павличин и Цачи Вайсман, знакомы широкой аудитории, поскольку оба были консультантами телешоу «Силиконовая долина». Они консультировали по вопросам изобретения алгоритма сжатия, который должен был исследовать стартап. На самом деле оба они пытаются сделать нечто подобное в реальной жизни, но с упором на геномику.
Сначала нам нужно понять масштаб проблемы. Геномное секвенирование человека генерирует от десятков до сотен гигабайт данных. С этим можно справиться, но по мере удешевления секвенирования генов становится возможным проводить секвенирование всей популяции, и не один раз, а несколько раз, чтобы посмотреть, как меняется ситуация. Речь идет об эксабайтах данных и необходимости их обработки.
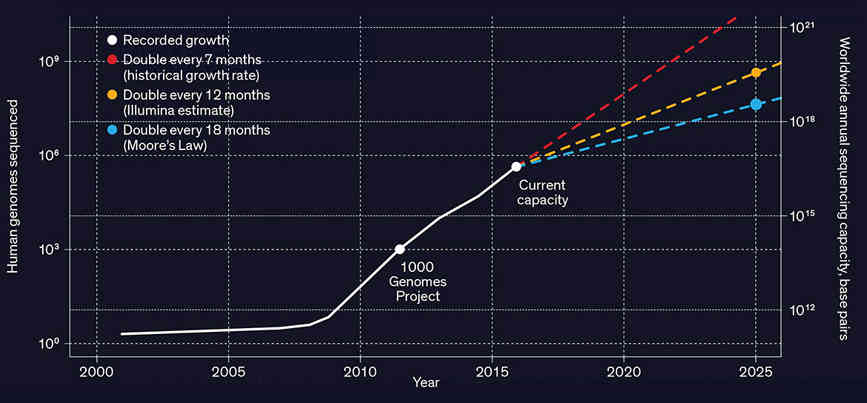
Необходим алгоритм сжатия, адаптированный к геномным данным. Проблема усугубляется тем, что данные очень фрагментированы. Машина для секвенирования выдает не длинную строку ACGT, кодирующую один геном, а большой набор перекрывающихся фрагментов, которые необходимо собрать. Данные также содержат ошибки, и секвенирующие машины выдают оценку качества, чтобы показать, насколько они уверены в данных. Все эти фрагменты и оценки качества выводятся в одном файле FASTQ, размер которого достигает 100 гигабайт.
Хорошая новость заключается в том, что, хотя данных много, в них много избыточности из-за повторного секвенирования определенных участков ДНК для проверки на наличие ошибок. Можно использовать общий алгоритм сжатия, например, ZIP или одну из последних модификаций словарного сжатия, но кажется более вероятным, что специфический для генома метод подойдет для требуемой обработки.
Например, один из подходов состоит в том, чтобы использовать одну последовательность генома в качестве эталонного словаря, а затем описать имеющиеся у вас последовательности с точки зрения начальной позиции и любых отклонений от стандарта. Это звучит хорошо, но это не имеет отношения к оценке качества.
Также предстоит работа по определению статистических характеристик ДНК. Если вы помните, все сжатие основывается на использовании вероятностных моделей, которые уменьшают энтропию данных от несжимаемых случайных данных к чему-то более регулярному и сжимаемому. Авторы статьи сообщают о найденном ими законе двойной силы, который придает определенную регулярность дисперсии генетических вариаций. Это интересное открытие само по себе с точки зрения того, какой аспект эволюции его вызывает, но оно также может быть использовано для точной настройки алгоритмов сжатия.
В настоящее время все алгоритмы сжатия генома работают без потерь, и вопрос о том, могут ли алгоритмы с потерями быть достаточно хорошими, остается открытым — не столько для последовательности ДНК, сколько для оценок качества, для хранения которых требуется много бит.
Это та область, где нам нужны новые идеи и новые способы обработки растущей горы данных.
Очевидно, что стандарты помогли бы будущей работе, и странным фактом является то, что Экспертная группа по движущимся изображениям — MPEG — является органом, работающим над созданием стандарта. Работа над MPEG-G должна быть завершена до конца этого года.
Последнее слово должно остаться за авторами:
Исследователи генома уже начали применять глубокое обучение к своим данным, но им, вероятно, придется подождать, пока накопится критическая масса геномной информации, прежде чем они смогут добиться подобных результатов. Однако ясно одно: Они не смогут достичь этого без серьезных достижений в технологии сжатия геномных данных».
